A revolução genómica foi possível graças às tecnologias de sequenciação conhecidas como de ‘nova geração’ ou de ‘alto rendimento’, que estão esboçadas no artigo recente ‘A que é que nos referimos quando falamos de revolução genómica?’ Os princípios da selecção genómica são:
- O ADN é analisado através de um chip para identificar diferenças no código genético entre animais, chamadas polimorfismos de nucleótido único (“Single Nucleotide Polymorphisms”, SNP).
- Os SNP's relacionam segmentos específicos de ADN com diferenças produtivas para determinados caractéres numa população concreta.


Um dos potenciais a longo prazo da selecção genómica é a identificação de genes epecíficos ou regiões genómicas com um impacto significativo na saúde suína e que permita melhorar a selecção para resistência e/ou tolerância a doenças. Alguns grandes projectos e colaborações de investigação estão a trabalhar nestas áreas e os seus resultados vão alargando a base de dados existente na literatura científica.
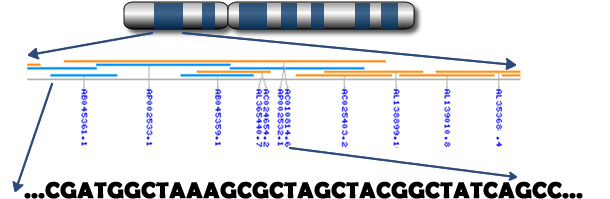
Figura 2. A genómica identifica SNPs para caractéres de saúde (Fonte: Prof. C. Haley – Roslin Institute, Edinburgh)
Por exemplo, numa edição da influente PAG (International Plant and Animal Genomics) no início de 2013, entre os principais artigos apresentados estavam os seguintes:
- Investigadores dos EUA identificaram três marcadores que estão significativamente associados com lesões pumonares causadas por pleuropneumonia e/ou pneumonia enzoótica. Espera-se que isto permita identificar a variação genética relacionada com o aumento da resistência às doenças respiratórias.
- Cientístas chineses investigaram os mecanismos que explicam a diferença no status sanitário entre porcos Landrace (susceptíveis) e Tongcheng (uma raça chinesa, que é resistente à exposição à PRRSV). Observaram a expressão diferenciada do gene CD169 nos porcos Tongcheng em comparação com os Landrace após a infecção por PRRSv. Alguns estudos apresentados mostram que o receptor CD169 melhora a apresentação do antigénio às célulasT.
- O PRRS Host Genetics Consortium (PHGC) foi criado por grupos de investigação do Canadá e EUA para determinar o papel da genética na resistência à infecção por PRRSv e os efeitos relacionados com o crescimento. Os últimos resultados de estudos em leitões em transição identificaram uma região genómica no cromossoma 4 (SSC4) com um impacto significativo sobre a variabilidade da carga viral e o crescimento após a exposição ao vírus. Análises posteriores e a sequenciação da região SSC4 devem identificar marcadores que permitam diferenciar entre animais resistentes a PRRSV/com crescimento máximo dos susceptíveis a PRRSV/com crescimento reduzido.
- Outros projectos do PHGC estudam o transcriptoma dos porcos expostos a PRRSv. Para que os genes produzam proteínas, o ADN transcreve-se para RNA mensageiro (mRNA). O termo transcriptoma inclui todo o mRNA que utilizado para a produção de proteínas. Este estudo proporcionará mais dados para decifrar os mecanismos genéticos da resposta do hospedeiro frente à infecção por PRRSv.
- Investigadores de Iowa State apresentaram uma análise genómica que revelou diferenças de expressão em muitos genes entre porcos classificados como excretores de salmonela persistentes ou moderados. Concluiram que as diferenças quantitativas nos valores de IFN-γ explicam a expressão da maioria dos genes analisados e que o regulão IFN-γ é uma fonte de genes cujo nível de expressão dos dias pós-infecção pode prever a capacidade excretora do animal. Estes genes podem ser avaliados como candidatos para o desenvolvimento de análises sobre a capacidade excretora dos animais.
- Investigadores chineses estudaram a resposta imunitária em leitões para 18 caractéres hematológicos, 7 linfocitários e 3 das citoquinas utilizando uma vacina contra peste suína em dois genótipos diferentes. Identificaram 12 SNPs significativos correlacionados com a resposta imune, o que sugere uma base preliminar para uma posterior identificação das mutações causais subjacentes à resposta imunitária geral.
- Investigadores de Nebraska trabalharam com raças cruzadas infectadas experimentalmente com PCV2b para identificar as principais variações genéticas que influem sobre os principais indicadores da progressão da doença e a resposta imunitária frente a PCVAD. Durante um periodo de 28 dias de exposição ao vírus, foi medido semanalmente o GMD, a carga viral e os valores de anticorpos específicos. Os resultados iniciais mostraram variação entre hospedeiros relacionados com a magnitude e tempo de resposta imunitária frente a PCV2b. Os principais clústers de SNPs que afectaram a carga viral estavam localizados em múltiplos cromossomas incluindo SSC6, SSC7 e SSC12. A região localizada em SSC7 também afectou o ganho de peso durante a exposição. O clúster de genes para o antigénio leucocitário suíno classe II localiza-se em dita região e pode explicar grande parte da variação fenotípica observada.

Figura 3. PCVAD – um objectivo chave futuro para a selecção genómica (Foto: J. Mackinnon).
Além dos estudos citados, estão a ser investigados os genes/SNPs envolvidos em outras doenças relevantes como a Febre Aftosa, a PSA e a Gripe Suína. No entanto, a selecção futura para uma melhoria na saúde requerirá o uso de múltiplos marcadores e a disponibilidade de grandes bases de dados de ADN de várias populações bem caracterizadas tanto genética como fenotipicamente nas que possam ser identificadas as associações entre SNPs e caractéres de saúde. O próximo artígo desta série, ‘Quando dará fruto a genómica?’, analisará isto com mais detalhe.