Os sinais clínicos da infecção pelo virus PRRS (PRRSV) podem ser diversos, pelo que devem ser confirmados mediante análises laboratoriais. Os mais frequentes são ELISA e RT-PCR, que detectam anticorpos específicos para PRRSV ou ácido nucleico viral, respectivamente.
A eleição do método de diagnóstico mais adequado depende do objectivo do diagnóstico. Para monitorizar explorações negativas preferem-se métodos muito específicos, enquanto que para analisar sémen de varrascos individuamente são requeridos métodos de máxima sensibilidade e que detectem a infecção na fase o mais precoce possível. Ao planear e executar um programa de controlo de PRRS, o melhor é optar por uma combinação de ELISA e PCR seguidos pela sequenciação de DNA. O protocolo de amostragem numa exploração concreta deve planificar-se ex professo, dependendo do objectivo do diagnóstico e da organização da exploração. A pergunta mais simples, se uma exploração está ou não infectada por PRRSV, pode ser respondida com um teste ELISA a 10-20 porcos de engorda. Uma análise detalhada da circulação de PRRSV numa exploração de ciclo fechado ou numa multisítio, requer a análise de um elevado número de amostras que representem vários grupos etários e de todos os sítios, já que podem representar ecossistemas virais independentes.

A interpretação da seroconversão ou da detecção do vírus em explorações onde se utilizam vacinas vivas modificadas representa um desafio já que (1) não há vacinas marcadas no mercado, (2) os vírus vacinais podem persistir nas explorações vacinadas e (3) pelo menos durante um tempo podem coexistir com as estirpes selvagens do vírus. Nas referidas explorações, é necessária a sequenciação de fragmentos de DNA obtidos em distintos grupos etários para entender totalmente a circulação do vírus.
A análise serológica mediante ELISA é fácil de levar a cabo e costuma ter uma boa especificidade e sensibilidade quando se aplica a toda a exploração. Contudo, o soro de animais individuais, especialmente de porcos adultos, pode produzir falsos positivos. Isto é um problema na monitorização de populações negativas e requer a repetição da análise das amostras com um método distinto (p.e.: IPMA, IFA ou outro teste ELISA de maior especificidade), ou uma nova recolha de amostras 2–3 semanas depois. Esta parte do rendimento dos testes ELISA raramente se tem em conta quando se avaliam as competências diagnósticas. A baixa sensibilidade de um teste ELISA concreto pode-se compensar aumentando o número de amostras analisadas, enquanto que a baixa especificidade requer sempre a aplicação de ferramentas adicionais.
Geralmente, qualquer teste ELISA é útil para a detecção de anticorpos contra qualquer genótipo de PRRSV, mas os kits que só utilizam antigenes de um genótipo costumam ser menos sensíveis contra outro genótipo. Também há kits para discriminar a seroconversão entre os dois genótipos mas, devido à reacção serológica cruzada entre ambos, os resultados devem interpretar-se com cuidado. É sempre recomendável a discriminação com PCR para confirmar o diagnóstico da co-infecção de uma exploração com ambos genótipos.
Os métodos PCR detectam ácidos nucleicos virais baseando-se na união complementar de oligonucleótidos sintéticos curtos (primers) a um fragmento concreto do genoma viral, que é amplificado posteriormente mediante uma reacção enzimática. Na maioria dos métodos PCR em tempo real o produto da reacção detecta-se mediante a monitorização do sinal emitido pela sonda DNA após se unir à sequência específica. Devido à natureza desta reacção, este sinal também pode ser emitido na ausência da sequência viral, especialmente nos últimos ciclos da PCR, o que pode parecer um positivo débil.


Ellingson 2013, thesis, Iowa SU
Contudo, a maior preocupação relativamente aos métodos RT-PCR está relacionada com a elevada diversidade genética do vírus o que pode dar lugar à acumulação de mutações no nucleótido dentro dos fragmentos de genoma nos que se unem os primers e as sondas pelo que podem dar falsos negativos da PCR. Uma análise comparativa de kits comerciais RT-PCR levada a cabo por um laboratório de referência da OIE (Podgorska et al., 2015) indicou que alguns dos kits falharam perante muitas estirpes tipo 1 do PRRSV originárias maioritariamente do Leste da Europa. Devido à limitada disponibilidade de sequências da Europa de Leste, a maioria dos primers e sondas utilizadas nos métodos actuais desenvolvem-se com base nas estirpes clássicas de tipo 2 e tipo 1 subtipo 1, enquanto que as estirpes da Europa de Leste dos subtipos 2-4 são geneticamente muito divergentes (Stadejek et al. 2013). Além disso, algumas delas demostraram ser mais virulentas que as estirpes circulantes no Ocidente e Centro da Europa, pelo que se lhes deveria prestar uma atenção especial para assegurar uma detecção precode no caso da sua propagação.
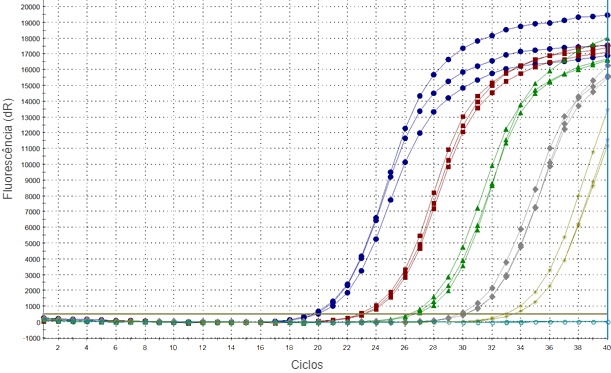
Actualmente, não se pode recomendar nenhuma análise RT-PCR como método universal que permita a detecção de todas as estirpes de PRRSV com uma sensibilidade óptima. Estas observações põem em relevo a necessidade de uma avaliação exaustiva dos métodos utilizados na actualidade e a sua validação constante em função das estirpes circulantes em cada momento. Para isso, deve-se criar uma colecção de estirpes de PRRSV que abarquem a diversidade genética conhecida do vírus, o que neste momento é um dos objectivos mais importantes dos laboratórios de referência de PRRS.








