E. coli (EC) – cultura e serotipagem
A E. coli é uma bactéria gram-negativa em forma de bacilo que tem uma multiplicidade de variantes. Além de estirpes comensais, que de maneira natural se encontram na flora intestinal de todos os mamíferos, também existem estirpes patogénicas classificadas como E. coli intestinal (InPEC) ou extraintestinal (ExPEC). No entanto, a diferenciação destas variantes vem colocando, há muito tempo, um problema de diagnóstico devido ao facto de que diferentes patotipos ou patovares não podem ser identificados com métodos fenotípicos simples.

A E. coli pode ser detectada nos porcos com relativa facilidade através da cultura em agar sangue (figura 1) ou meios diferenciais (p. ex., agar Gassner ou MacConkey) de amostras adequadas (intestino delgado, fezes) de animais com infecção aguda. Há alguns anos, foi utilizado com êxito a tipificação de serótipos ou serovares para diferenciar isolados de E. coli, devido ao facto de que certos serótipos estão associados frequentemente a determinadas doenças (tabela 1). Por isso, em veterinária, é habitual determinar os antigénios O e F, por aglutinação rápida em placa utilizando soros poli- ou monovalentes (os antigénios O são componentes dos LPS da parede celular bacteriana e são também endotóxicos; os antigénios F [fimbrial] são adesinas que facilitam a adesão hóspede-específica para colonizar as células epiteliais do intestino delgado). Ainda que este método seja rápido, está sujeito a erros por reacções cruzadas, ocultação de antigénios diana ou incapacidade das fimbrias para se desenvolverem in vitro, o que pode conduzir a classificações erradas. Por outro lado, desde há décadas que a serofórmula O:K:H tem sido e continua a ser o standart para uma determinação completa de todos os serótipos, p. ex., para determinar não só os antigénios O, mas também os K (polissacárido capsular) e H (proteína flagelar).
Tabela 1: Patogénios E. coli intestinais em porcos
| Patótipos |
Doenças causadas por E. coli |
Serótipos de antigénio O frequentemente associados | Serótipos fimbriais frequentemente associados |
| E. coli patogénio intestinal (InPEC) | Diarreia por E. coli em leitões | O8, O108, O138, O139, O141, O147, O149 e O157 | F4, F5, F6 e F41 |
| Diarreia por E. coli em porcos desmamados | O8, O108, O138, O139, O141, O147, O149 e O157 | F4, F18ac | |
| Doença dos edemas em porcos desmamados | O138, O139 e O141 (O147, O157 e rotineiramente não serotificáveis) | F18ab |
E. coli (EC) – patótipos e virotipificação
Outros avanços nos métodos de biologia molecular e melhorias na análise biológica molecular da E. coli levaram à identificação de muitos genes que codificam factores de virulência (tabela 2): por um lado, factores de virulência clássicos com efeitos nocivos (toxinas) e, por outro, aqueles que directa ou indirectamente facilitam a colonização e a sobrevivência da E. coli no hospedeiro. A identificação destes genes, por exemplo, por PCR ou técnica array, faz com que actualmente seja possível determinar a virulência potencial de isolados de E. coli e distinguir entre diferentes patótipos (tabela 3). A identificação destes marcadores de virulência e, portanto, dos seus genes diana é chamada de virotipificação.
Tabela 2: Exemplos de genes diana detectados em E. coli e os factores de virulência
(fímbrias, adesinas, toxinas) para os que codificam
| Genes diana | Factores de virulência |
| faeG (F4) | Fímbrias F4 |
| fanC (F5) | Fímbrias F5 |
| fasA (F6) | Fímbrias F6 |
| fedA (F18) | Fímbrias F18 |
| fim41A (F41) | Fímbrias F18 |
| fimH (F1) | Fímbrias tipo 1 |
| fimA (F1) | Fímbrias tipo 1 |
| papC | Fímbrias P |
| aidA (AIDA) | Adesina autotransportadora AIDA-I |
| paa | Factor de aderência suíno |
| eaeA (intimina) | Intimina |
| eltB (LTI) | Enterotoxina sensível ao calor |
| estA (STI) | Enterotoxina resistente ao calor |
| est B (STII) | Enterotoxina resistente ao calor |
| estA (EAST) | Enterotoxina resistente ao calor |
| stx2e | Shigatoxina variante 2e |
| cdtB | Toxina citoletal distensora |
| cnf1 | factor citotóxico necrotizante tipo 1 |
| iucD | Aerobactina |
| escV | Sistema de secreção tipo III |
| pic | Autotransportador serin protease |
Tabela 3: Genes diana para a identificação de patótipos de E. coli em porcos
| Patótipos | Fímbrias e adesinas (genes diana) | Toxinas (genes diana) | Sintomas | |
| E. coli patogénico intestinal (InPEC) |
E. coli enteropatogénica (EPEC) |
fimA/fimH paa intimina |
escV pic cdtB |
diarreia por má absorção |
|
E. coli enterotóxica (ETEC)  |
F4, F5, F6, F18, F41 fimA/fimH AIDA paa |
STI, STII, EAST LTI Stx2e hemolisina |
diarreia por hipersecreção | |
| Doença dos edemas por E. coli (EDEC) | F18 AIDA |
Stx2e hemolisina |
doença dos edemas | |
| E. coli formadora de Shigatoxina (STEC) | fimA, fimH intimina |
Stx2e EAST escV pic, cdtB hemolisina |
diarreia catarral-hemorrágica | |
| E. coli patogénico extraintestinal(ExPEC) |
E. coli septicémica (SEPEC) |
papC | iucD | septicémia hemorrágica |
| E. coli uropatogénica (UPEC) | papC fimA, fimH | cnf1, iucD hemolisina |
mastite por coliformes em porcas | |
Não obstante, os isolados de E. coli patogénico contém sempre uma combinação de fímbrias e adesinas, além de toxinas. Várias toxinas e adesinas podem estar presentes ao mesmo tempo, e em diferentes combinações. A determinação destes diversos marcadores de virulência e a identificação dos seus genes proporciona informação sobre o patótipo de E. coli isolado da cultura celular (tabela 3). Há que destacar que os E. coli hemolizantes não são os únicos que são patogénicos (figura 1) e, de facto, os E. coli não hemolizantes, em particular, são determinados com frequência como causa de doença clínica. Por outro lado, os E. coli hemolizantes não costumam possuir genes relevantes para fímbrias e toxinas. Portanto, se for possível, todas as variantes de isolados de E. coli identificadas por cultura devem ser virotipificadas com o objectivo de assegurar que o agente patogénico responsável possa também ser identificado. A informação sobre os tipos e toxinas fimbriais de E. coli patogénico determinados também é útil na selecção de vacinas adequadas com os antigénios correspondentes.
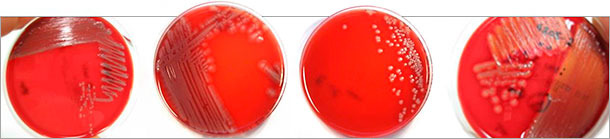
Figura 1: Diferentes morfologias coloniais de isolados de E. coli patogénicos em agar sangue (Columbia) identificados após virotipificação por PCR múltiplo. Não apenas os E. coli hemolizantes (direita) são patogénicos.
Na interpretação dos resultados de diagnóstico há que ter em conta que os patótipos de E. Coli também podem ser isolados em hóspedes sãos (infecção latente), sobretudo da flora intestinal. Portanto, a avaliação dos resultados só pode ser feita tendo em conta tanto as descobertas clínicas no animal como a concentração dos agentes patogénicos isolados com o patótipo identificado.
A interpretação do virotipificado torna-se complicada quando os marcadores de virulência são detectados directamente no material da amostra, p. ex. fezes, sem necessidade de tipificar os isolados individuais. Ainda que isto seja tecnicamente possível e realizado sob pedido, quando diferentes patótipos de E. coli estão presentes na amostra, também se obtém uma mistura de todos os factores detectáveis, que pode conduzir à identificação de falsos patótipos e uma combinação de marcadores de virulência que não existe como tal. Além disso, também é possível que sejam detectados marcadores de virulência de outras bactérias intestinais presentes na amostra de fezes, já que estas bactérias têm genes semelhantes. Neste caso, serão detectados possíveis marcadores de virulência de E. coli que, de facto, não são factores de virulência para a E. coli detectado na amostra. Portanto, deve ser tido um especial cuidado na interpretação dos resultados obtidos na virotipificação das amostras.
Em combinação com o exame clínico e o frequentemente necessário exame patológico (descrito anteriormente) e com diagnósticos diferenciais apropriados, o virotipificado dos isolados de E. coli proporciona uma notável melhoria no diagnóstico das infecções por E. coli no porco.



