A diversidade dos vírus da gripe pode ser explicada principalmente por dois aspectos muito relacionados entre si. Por um lado, os mecanismos evolutivos principais a que este vírus está submetido — a deriva e a reordenação ou intercâmbio genético (discutidos em outro artigo desta mesma secção)— por outro, o comportamento epidemiológico do vírus em cada espécie, o qual, como será discutido no presente artigo, tem um efeito importante sobre os mecanismos evolutivos.
Se, com base na deriva genética, forem comparados os vírus presentes no porco com os presentes no ser humano, há a destacar principalmente dois fenómenos: 1) dada uma estirpe concreta, o vírus no ser humano varia antigenicamente (mudanças nos aminoácidos) mais depressa que no porco, ainda que a taxa de mutação genética do vírus seja igual para ambas as espécies e 2) a nível global, a diversidade do vírus da gripe, tanto a nível genético como antigénico, é muito maior em porcos que no ser humano. Apesar destes dois factos poderem parecer a priori contradictórios, são, como se verá mais à frente, perfeitamente compatíveis.

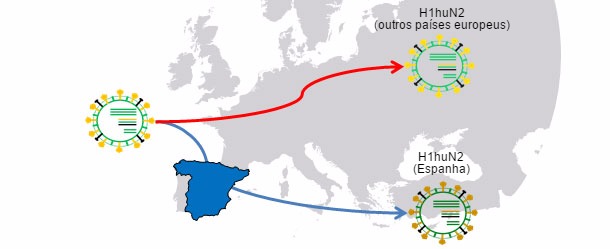
Figura 1. Ancestral común H1huN2 (1994); Isolamento geográfico → a deriva produz variantes relacionadas com cada país.
Em outros artigos desta mesma secção vimos que a gripe no porco pode comportar-se de forma endémica ou enzoótica ou, dito de outro modo, que os vírus da gripe podem "residir" nas explorações. Esta situação é muito frequente e leva ao aparecimento de circulações recorrentes. Num trabalho do ano 2001 (de Jong e colaboradores) compararam-se genética e antigenicamente mais de 20 H1N1 suínos de "tipo aviário" (H1avN1). Estes vírus tinham sido isolados em 6 explorações da Holanda que estavam endemicamente infectadas. Com base no gene da hemaglutinina (H), comprovou-se que os isolados formavam cinco grupos que se diferenciavam tanto a nível genético como antigénico. Há a destacar que cada grupo correspondia a uma exploração (salvo um dos grupos que incluia isolados de duas explorações). Este trabalho demonstra que todos os isolados têm um H1avN1 ancestral comum mas que, graças à circulação endémica, os vírus evoluiram e diferenciam-se de forma isolada. Noutro estudo (Martín-Valls et al., 2013), realizado no CReSA entre os anos 2009 e 2012, isolaram-se 4 vírus H1N2. Ao analisar a H1 de um ponto de vista genético, observou-se que todos eles pertenciam ao tipo H1N2 "tipo humano" (H1huN2), mas que formavam um grupo claramente diferenciado dentro dos H1huN2; poderia concluir-se que tinha existido uma evolução isolada em Espanha deste tipo. Posteriormente, tem sido possível comprovar que estes vírus reagem de forma limitada com soros que reagiam com outro H1huN2 comercial, indicando certa deriva antigénica (dados não publicados). Definitivamente, tanto os factores epidemiológicos como a evolução por isolamento em explorações em situações endémicas, ou o isolamento geográfico, jogam um papel importante no aparecimento de novas variantes antigénicas. No ser humano, no entanto, a situação é completamente diferente. Neste caso pode-se dizer que, devido à pressão imunitária global, os vírus são forçados a mudar antigenicamente para poder sobreviver. Actualmente, as múltiplas e variadas conexões que existem entre países de todo o mundo eliminam as situações de isolamento. Portanto, no ser humano existe uma selecção positiva global daquelas variantes de vírus de gripe capazes de escapar da resposta imunitária, este fenómeno traduz-se na necessidade constante de uma actualização das vacinas no ser humano.
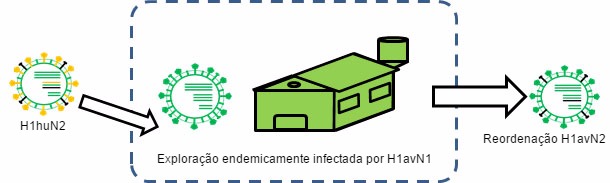
Figura 2. H1huN2 é transmitido a uma exploração endemicamente infectada por H1avN1 → Produz-se reordenação.
A reordenação ou intercâmbio genético também pode ser origem de mudanças a nível antigénico, inclusive de uma forma mais súbita. No ser humano, por exemplo, esta foi a causa principal das últimas 3 pandemias já que levou à introdução de, pelo menos, uma nova hemaglutinina diferente antigenicamente da pré-existente na população e contra a qual não havia imunidade anterior. Em outro artigo desta secção já vimos como, no porco, os vírus da gripe suína sofrem reordenações de forma frequente, gerando-se, em consequência, novas estirpes. Exemplos disso são o H1avN2, um H1N2 que cruza antigenicamente com os vírus H1avN1 (Rose et al., 2013), ou os H1huN1, que cruzam antigenicamente com o H1huN2 (Martín-Valls et al., 2013; Rose et al., 2013). Para que estas reordenações ocorram é necessário que duas estirpes diferentes infectem um mesmo animal ao mesmo tempo. Obviamente, as situações endémicas são cenários ideais para que isto aconteça. De facto, quando as situações endémicas foram avaliadas, foram observadas evidências de reordenação genética entre diferentes estirpes e inclusive subtipos (Martín-Valls et al., 2013; Rose et al., 2013).
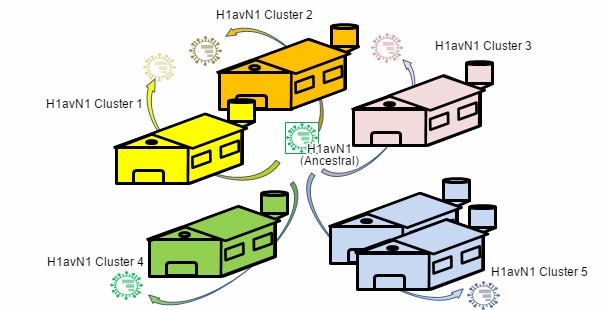
Figura 3. As infecções endémicas facilitam a evolução isolada por deriva em cada exploração→ heterogenicidade genética e antigénica relacionada com a exploração.
Em conclusão, os vírus da gripe suína apresentam uma alta diversidade genética e antigénica gerada pelos mecanismos evolutivos intrínsecos dos vírus. Dita diversidade é potenciada por factores epidemiológicos como as situações de circulação endémica e/ou o isolamento geográfico, tendo-se acentuado ao longo do tempo. A interacção de todos estes factores pode ter implicações na falta de reconhecimento imunológico, ou seja, num deficit na protecção cruzada entre estirpes pertencentes a um mesmo subtipo.




