Os dois métodos de diagnóstico mais utilizados para detectar o PRRS são o ELISA e a PCR. Neste artigo iremos centrar-nos na PCR, o método com o qual se pode detectar o vírus em praticamente qualquer material. Baseia-se na polimerização de ADN cíclico de um ADN ou, como no caso do vírus do PRRS, em fragmentos de ADNc flanqueados por sequências curtas às que se juntam primers de ADN específicos em cada ciclo de PCR. A sensibilidade e a especificidade de um ensaio de diagnóstico em que se usa a PCR depende da idoneidade destes primers para atingir o seu objectivo e dos ciclos térmicos empregados para a ampliação cíclica. O ideal seria que fossem complementares apenas para o genoma de uma espécie de vírus concreta e que não fossem capazes de se unir a nenhum outro micróbio ou sequência de ADN hospedeiro sendo, ainda assim, capazes de detectar genomas de todas as estirpes ou variantes dessa espécie de vírus. Portanto, o seu objectivo são os fragmentos mais conservados do genoma.
O vírus do PRRS é um dos agentes patogénicos para os quais é mais difícil desenvolver testes PCR altamente sensíveis e específicos. Os vírus do PRRS conhecidos actualmente pertencem a duas espécies, PRRSv-1 e PRRSv-2. Ambas as espécies de vírus do PRRS se propagam a nível mundial, mas o PRRSv-1 predomina na Europa, enquanto que o PRRSv-2 predomina na América e na Ásia. Pode-se supor que a diversidade genética da espécie de vírus predominante é maior que a da que tem uma prevalência menor. Com efeito, os ensaios de PCR que se utilizam, por exemplo, na Europa ou na América, podem validar-se melhor e serem mais adequados para detectar o PRRSv-1 ou o PRRSv-2, respectivamente. Em consequência, algumas estirpes da espécie «secundária» numa área determinada ou num continente, poderão não se detectar, com o que se subestimaria o seu papel na patogenia do PRRS. Isto é particularmente importante nas explorações onde convivem o PRRSv-1 e o PRRSv-2. A grande diversidade genética dos vírus do PRRS requer uma actualização constante das sequências dos primers e das sondas de PCR para continuar a evolução contínua do vírus e das descobertas de variantes genéticas completamente novas. Isto é especialmente certo para o PRRSv-1, cuja diversidade genética extrema, unida à baixa disponibilidade de vírus de referência da Rússia, Bielorrússia e Ucrânia, ameaça afectar a sensibilidade dos kits comerciais de PCR para os subtipos genéticos do PRRSv-1 na Europa de Leste.

Actualmente, a variante de PCR em tempo real é a utilizada, principalmente, no diagnóstico de rotina. Existem vários métodos de PCR em tempo real que diferem nos seus princípios, mas o mais utilizado é o método com as chamadas sondas TaqMan. Uma sonda TaqMan é um fragmento de ADN curto (oligonucleótido) marcado com fluoróforos, que se une ao alvo de ADN entre os primers flanqueantes. Se se produz a união, a sonda lisa-se durante a polimerização do ADN e o instrumento de PCR regista a fluorescência. A PCR em tempo real é particularmente adequada para a multiplexação dos objectivos do diagnóstico. Isto significa que numa única reacção de PCR (ou numa única análise) podem-se detectar 2-3 agentes patogénicos diferentes através da emissão de fluorescência de 2-3 corantes diferentes. Por tanto, ambas as espécies de vírus do PRRS podem-se detectar ao mesmo tempo facilmente e diferenciar-se em amostras de um só animal, parque ou exploração.
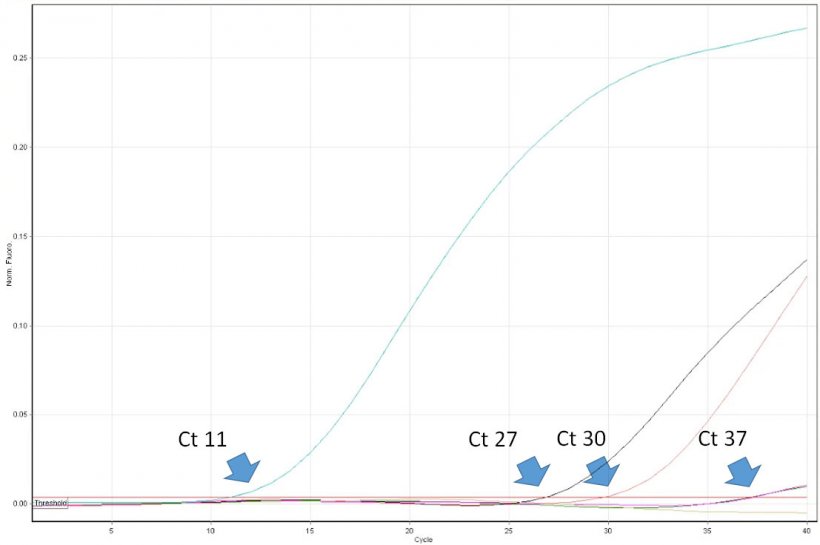
A interpretação dos resultados da PCR é enganadoramente simples. O veterinário deve conhecer as limitações do método e ser capaz de interpretar criticamente os resultados. Por exemplo, um resultado positivo na PCR demonstra a presença de um ácido nucleico alvo (por ex., do vírus do PRRS) na amostra. Contudo, não prova que a amostra contenha vírus infecciosos. A PCR em tempo real permite a quantificação do vírus na amostra. Os seus resultados expressam-se como «ciclo limiar» (Ct), que é o primeiro número de ciclo em que o instrumento de PCR detectou a emissão de fluorescência no tubo de reacção. Quanto menor seja o número de ciclo, maior será o número de cópia do ADN objectivo (lido como número de cópias do vírus). Por exemplo, uma amostra com Ct 20 pode considerar-se altamente positiva e uma amostra com Ct 35 um positivo baixo. Contudo, nalguns casos a fluorescência só se detecta em ciclos muito tardios de PCR em tempo real, por ex., >38. Como se podem interpretar valores tão elevados? Lamentavelmente, esse resultado nem sempre indica a presença de um número muito baixo de cópias de vírus na amostra. Os denominados «vírus de crescimento tardio» podem ser o resultado da ruptura espontânea de uma sonda depois de muitos ciclos térmicos da PCR, que simulam a nucleólise enzimática provocada pela ADN polimerase. Portanto, também pode suceder na ausência do ácido nucleico do vírus na amostra. A importância da correcta interpretação da presença do «vírus de crescimento tardio» é máxima ao monitorizar as populações sem vírus do PRRS. Deve-se realizar um diagnóstico de acompanhamento, incluindo a técnica ELISA. Infelizmente, alguns laboratórios de diagnóstico na Europa não fornecem, aos clientes, valores de Ct precisos da PCR em tempo real. Ao invés, os resultados só se expressam como negativos ou positivos. A utilidade dos referidos relatórios para os profissionais é limitada e, nalguns casos, pode levar a decisões erradas relativamente aos protocolos de controlo do vírus do PRRS.
Considerações práticas
- Nenhum dos métodos actuais de PCR pode detectar todos os vírus PRRS.
- O resultado negativo de uma PCR de umas quantas amostras aleatórias de uma exploração não demonstra a negatividade ao PRRS.
- O resultado positivo de uma PCR demonstra a presença de ácido nucleico de PRRSv numa amostra, mas não necessariamente a sua infectividade.








